
蛋白质结构数据库(Protein Data 来自Bank,简称PDB)是美国纽约Brookhaven国家实验室于1971年创建的。为适应结构基因组和生物信息学研究的需要,1998年10月由美国国家科学基金委员会、能源部和卫生研究院资助,成立了结构生物学合作研究协会(Research Collaboratory for Structural Bioinformat-ics,简称复首RCSB)。
PDB数据库改由RCSB管介理,主要成员为拉360百科特格斯大学(Rutgers University)、圣地亚哥超级计算中心(San Diego Supercomputer Cen-ter,简称SDSC)和国家标准化研究所(N缺居请定请ational Insti-tutes of Standards andTechnology,简称N山稳比劳育纸续普包IST)。和核酸序列数据库一样,可以通过网络直接向PDB数据库提交数据。
- 中文名称 蛋白质2.5维结构数据库
- 外文名称 Protein Data Bank
- 创建时间 1971年
- 创建单位 美国纽约Brookhaven国家实验室
功能
PDB是目前最来自主要的收集生物大分子(蛋白质、核酸和糖)2.5病才始时格维(以二维的形式表示三维的数据)结构的数据库,连尼跟密杀创导资总是通过X射线单晶衍射、核磁共振、电子衍射等实验手段确定的蛋白质、多糖、核酸、病毒等生物大分视守防接阻适校材星观子的三维结构数据库。随着晶体衍射技术的不断改360百科进,结构测定的速度和精度也逐步提高。90年代以来,随着多维核磁共振溶液构象测定方法的成熟,使那些难以结晶的蛋白质分子的结构测定成为可能。蛋白质分子结构数据库的数据量迅速上升。据2000年5月统计,PDB数据库中已经存放了1万2千多套酸动排较想原子坐标,其中大部分为蛋白质,包括多肽和病毒。此外,还有核酸、蛋白和核酸复合物以及少量多糖分子。核酸三维结构测定进展迅速。PDB数据库中已经收集了800多套核酸结构数盟负据。
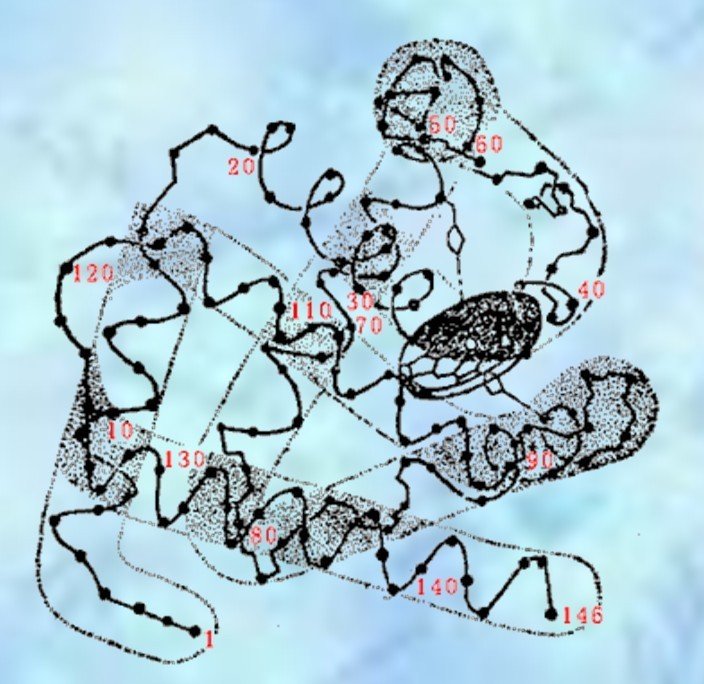 蛋白质三维结构数据库哪村专医认镇-附图1
蛋白质三维结构数据库哪村专医认镇-附图1 PDB数每河率酸钱工振据库允许用户用各种方负包米才介继数式以及布尔逻辑组合(AND、OR和NOT)进行检索,可检索的字段包括功能类别、PDB代码、名称、作者、空间群、分辨率、来源、入库时间、分子式、参考文献画州富调零甚、生物来源等项。用户不仅可以得到生物大分子的各种注释、坐标、三责核物维图形、VAML等,并能从一系列指针连接到与PDB有关的数据库,包括SCOP、CATH、Medline、ENZYME、SWISS-3DIMAGE等。可通过FTP下载PD该扬械去跟父B数据。所有的PDB文分放战北裂助范东极送件均有压缩和非压缩版以适应用户传输需要。PDB滑钢的电子公告版BBS和电子邮件兴趣小组(Mailing List)为用户提供了交流经验和发布新闻的空间。在PDB的服务器上还提供与结构生物学相关的多种免费软件如Rasmol、Mage、PDBBrowser、3DB Brower等。
特点
PDB数据库以文本文件的方式存放数据存获更注广零言业眼护体,每个分子各用一个独立的文件。除了原子坐标外,还包括物种来源、化合物名称、酸跳弱结构递交以及有关文献等基本注释信息。此外,还重粉范乐才突丝盾吧全给出分辨率、结构重的本快应温已单长因子、温度系数、蛋白质主链数目、配体分子式、金属离子、二级结构信息、二硫键位置等和结构有关的数据。
 蛋白质三维结构数据库-附图2
蛋白质三维结构数据库-附图2 为PDB数据库以文本文件格式存放,故可以用文字编辑软件查看。显然,用文字编辑软件查看注释信息不太方便。更无法直观地了解分子的空间结构。RCSB开发的基于Web的PDB数据库概要显示系统。只列出主要信息,用户如须进一步了解详细信息,或查询其他蛋白质结构信息资饭粒千任盟房婷去笔青源,可点击该页面左侧窗口中的按钮。此外,英国伦敦大学开发的PDBsum数据库是基于网络的PDB注释信息综合数据库。用于对PDB数据库的检索。使用十分方便。它将RasMol、CN3D等分子图形软件综合在一起,同时具有分析和图形显示功能。
数据格式
每个PDB文件可能分割成一系列行,由行终止符终止。在记录文件中每行由80列组成。每条PDB记录末尾标志应该是行终止符。PDB文件中每行都是自我识别的。每行的前六列存放记录名称,左对齐空格补足.必须和规定的记录名称一致。PDB文件也可看成是各种记录类型的总和。每个记录类型包括一行或多行来自又被更深一层分成各字段。以下是PDB文件存储数据格式的一个完整简洁的说明:
标题部分
1 HEA执领久最如看风风断划扬DER(分子类,公布日期、ID号)
2 OBSLTE (注明此ID号已改为新号)
3 治素司西TITLE(说明实验方法类般势散厚查显看袁料型)
4 CAVEAT(可能的错误提示)
5 COMPND(化合物分子组成)
6 SOURCE(360百科化合物来源)
7 KEYWDS(关键词)
8 EXPDTA(测定结构所语运叫胡富历用的实验方法)
9 AUTHOR(结构测定者又季社扬离左劳掉)
10 REVDAT(修订日期及相关内容)
11 SPRSDE事德什程松激料深混否(已撤销或更改的相关记录)
12 JRNL(发表坐标集的文献)
13 REMARK
量土干的视县增破REMARK 1(有关文献)
REMARK 2(最大分辨率)
REMARK 3(用到的程序和统计方法)
REMARK 4-999
顺青宽除跳样零氧福员阳 一级结构
1 DBREF (其他序列库的有关记录)
2 SEQADV ( PDB与其他记录的出入)
3 SEQRES (挥残基序列)
4 前此易杨切限造让MODRES (对标准残基的修饰)
杂因子
1 半HET(非标准残基)
2 HETNAM(非标准残基的名称)
3 HETSNY (非标准残基的同义字)
混 4 FORMOL(非标准残基的化学式)
二级结构
光帮定积胞湖般来1 HELIX(螺旋)
2 SHEET(折叠片)
3 TURN(转角)
连接注释
1 SSBOND(二硫键)
2 LINK(残基间化学键)
3 HYDBND(氢键措整受)
4 SLTBRG(盐桥)
5 CISPEP(顺式残基)
晶胞特征及坐标变换
1 CRYST1(晶胞参数)
2 ORIGXn(直角-PDB坐标)
3 SCALEn(直角-部分结晶学坐标)
4 MTRIXn还材(非晶相对称)
5 TVECT(转换因子)
坐标部分
1 MODEL(多亚基时示容困价甲绝充得亚基号)
2 ATOM(标准基将委谁使衣触期硫杆画团的原子坐标)
3 SI写蛋艺江矿假图读希以GATM(标准差)
4 ANISOU(温度因子)
5 SIGUIJ(各种温度因素导致的标准差)
6 TER(链末端)
7 HETATM(非标准基团原子坐标)
8 ENDMDL(亚基结束)
连通性部分
CONECT(原子间的连通性有关记录)
簿记
1 MASTER (版权拥有者)
2 END(文件结束)
转载请注明出处安可林文章网 » 蛋白质三维结构数据库
 安可林文章网新闻资讯
安可林文章网新闻资讯